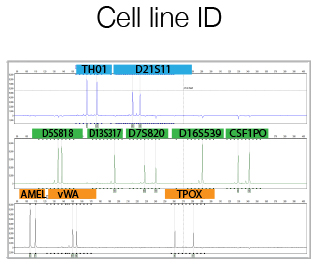
Cosa offriamo
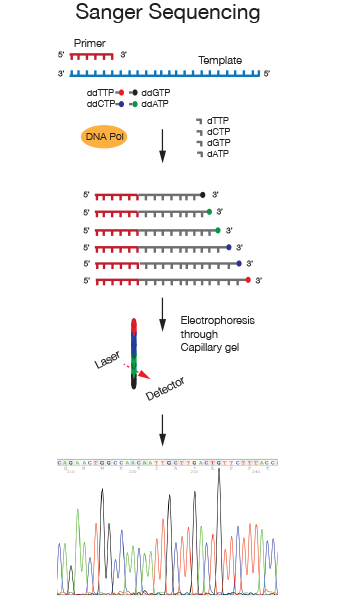
Il Servizio di DNA Sanger Sequencing nasce nei primi anni del 2000, con l’introduzione di strumentazione automatizzata a capillari e la chimica con terminatori fluorescinati BygDye, per garantire un'alta qualità di lettura a partire da diversi tipi di templato.
In più di 20 anni di esperienza in questo campo, abbiamo fornito supporto tecnico e scientifico a numerosi progetti di ricerca di base e traslazionale.
Oltre a ciò, il Servizio di DNA Sanger Sequencing collabora in prima linea all'attività di diagnostica del CGT lab.
Nello specifico, per il sequenziamento del DNA offriamo:
- le conservazione dei campioni fino ad 1 mese
- la disponibilità di primer universali gratuiti
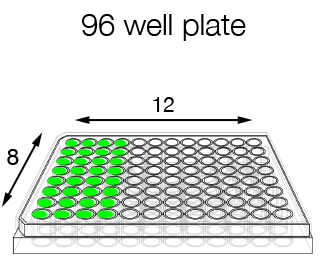
- la possibilità di lavorare in formato a 96 pozzetti (per campioni omogenei e da processare con lo stesso oligo), oltre che a tubo singolo
- il formato “Ready-to-go”, in cui il cliente fornisce il DNA mischiato al primer
- l’assistenza in qualunque fase del processo
- la consegna dei risultati in 24-48 ore
- le ricariche gratuite
- la spedizione dei dati in formato .ab1
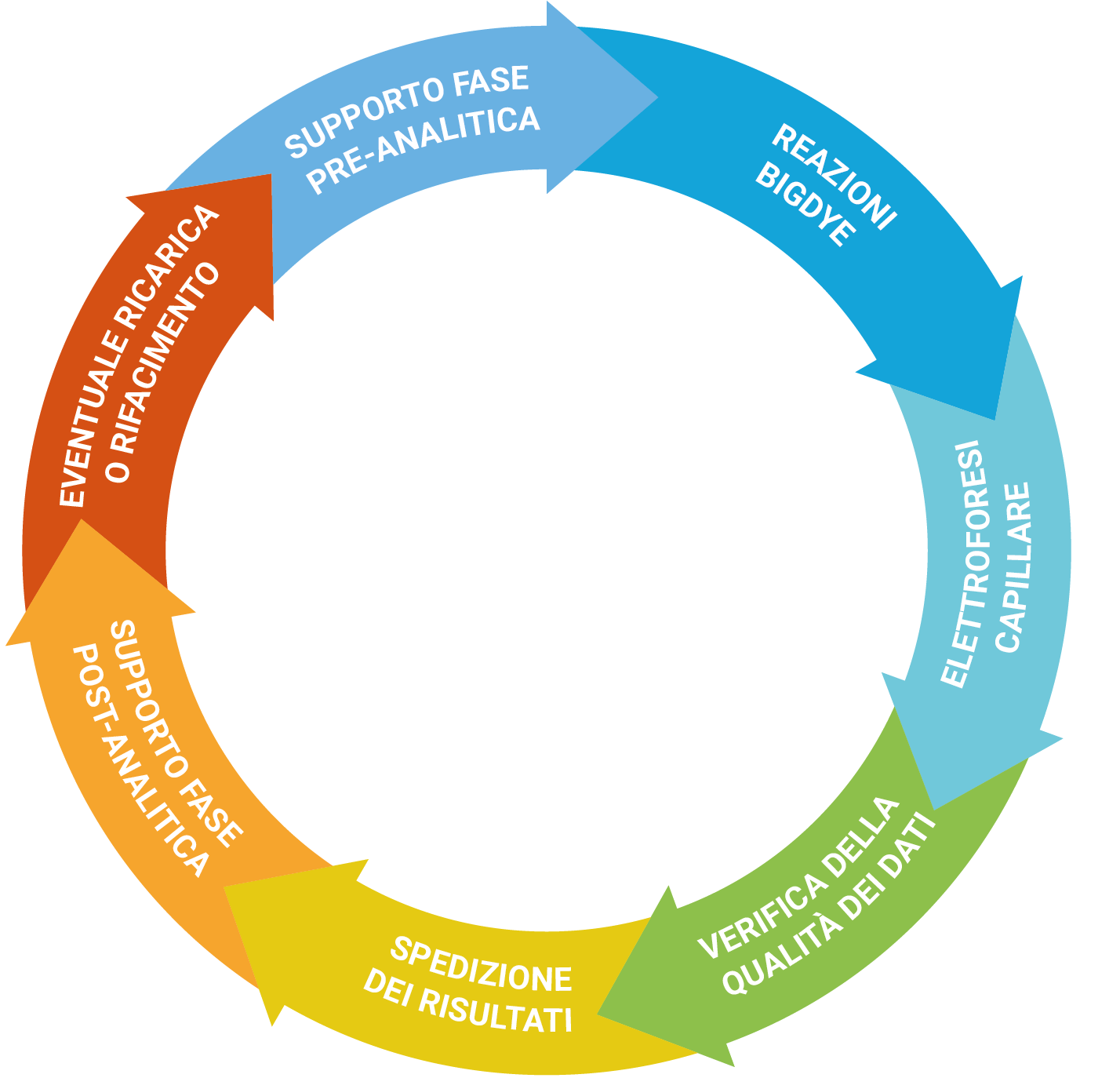
Servizio
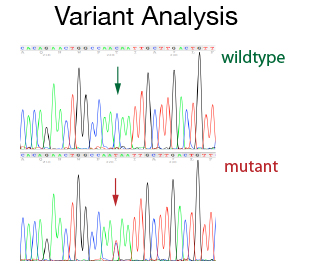
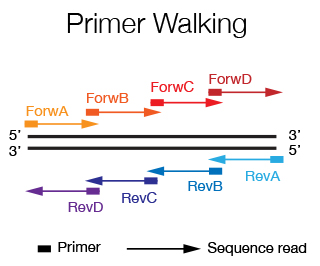
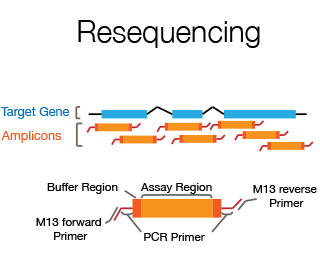
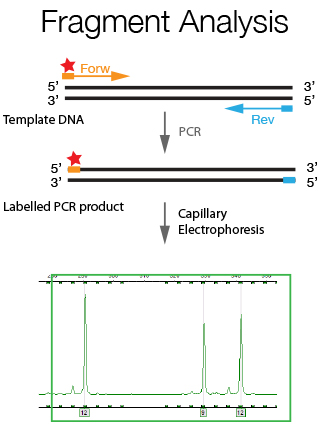
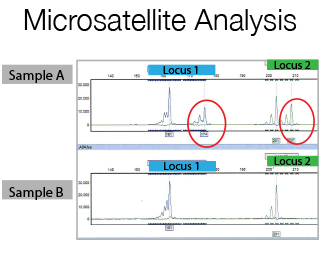
L'unità di DNA Sanger Sequencing è in grado di offrire il sequenziamento standard del DNA e lo sviluppo di progetti con metodo di Sanger. Offriamo la possibilità di utilizzare la tecnologia dell'analisi di frammenti per diverse applicazioni, fornendo assistenza nella fase pre-e post-analitica.
Innovazione
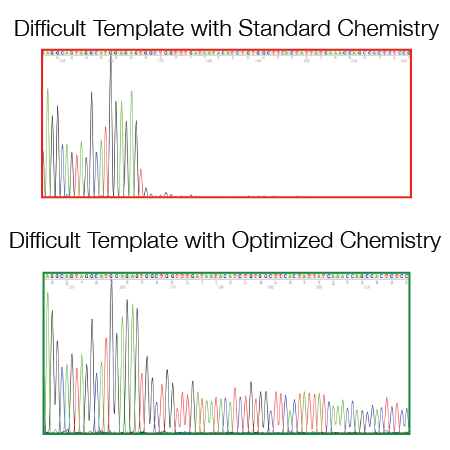
Lo Staff valuta il rinnovamento dei protocolli rispetto alle novità in campo tecnologico e di reagenti, per ottimizzare e migliorare il nostro lavoro e l'offerta fornita ai nostri utenti. Manteniamo una lista sempre aggiornata e ottimizzata di primers universali gratuiti.
Qualità
Il laboratorio è accreditato ISO 9001
Viene fornita una capacità di lettura di sequenziamento di 600-800 paia di basi, con accuratezza del 99.99% (Q 40).
UNI EN ISO 9001:2015 n. IT324391

Prestazioni
FAQ e Info Tecniche
Staff e Contatti

Sara Volorio
Sara Volorio si è laureata in Biologia nel 1995 presso l'Università di Milano. Ha lavorato per 5 anni all' Istituto TIGEM dove ha scoperto la sua passione per il sequenziamento. Si è quindi spostata nell' anno 2000 in IFOM dove è diventata successivamente la responsabile dell’Unità di sequenziamento del DNA con metodo Sanger, occupandosi anche dell’area R&D del CGT Lab.

Stefano Fortuzzi
Stefano Fortuzzi si è laureato nel 2005 in Biotecnologie Agrarie e Vegetali presso l'Università di Bologna. Negli anni successivi inizia a lavorare presso Cogentech nel gruppo di Test Genetici dove tuttora sviluppa ed attua l'automazione dei processi produttivi. Dal 2014 entra a fare parte dell'Unità di DNA Sanger Sequencing dove lavora tutt'oggi.

Domenico Sardella
Domenico Sardella nel 2003 ha conseguito il Diploma Universitario in Tecnico Sanitario di Laboratorio Biomedico presso l'Università degli Studi di Urbino. Dopo un'esperienza lavorativa presso l'Istituto Nazionale Tumori e l'Istituto Europeo di Oncologia, nel 2012 è stato assunto come Tecnico di laboratorio in Cogentech DNA Sanger Sequencing.
- Tel.
+39 02 574303210
+39 02.574303207 (lab) - Email
sequencing-service [@] cogentech.it